近日,我实验室董志国教授科研团队完成了青蛤全基因组图谱绘制,对埋栖适应的分子基础开展了深入研究并取得重大进展。研究成果以“Chromosome-level clam genome helps elucidate the molecular basis of adaptation to a buried lifestyle”为题发表在国际著名期刊《Cell》子刊《iScience》上。董志国教授和魏敏博士分别为本文的通讯作者和第一作者,实验室青年博士葛红星、中国水产科学研究院黄海水产研究所邵长伟、大连海洋大学闫喜武和聂洪涛为共同第一作者。该成果在江苏省优势学科海洋科学与技术和国家贝类产业体系等项目的共同资助下完成。该文章为我校首次以第一作者和通信作者单位在iScience上发表的研究论文,江苏海洋大学为论文唯一知识产权单位。
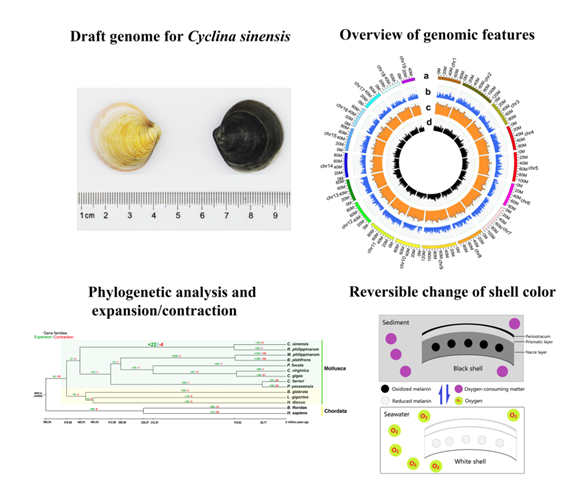
青蛤是我国重要的浅海滩涂贝类。过去由于青蛤基因组信息的匮乏成为其遗传选育的障碍。破译青蛤基因组信息、开展重要性状遗传改良成为当下的重要课题。该团队首次完成了高质量青蛤全基因组图谱的绘制,contig N50达到2.6 Mb。基因组大小903.2M,包含27,564个基因。本研究发现埋栖贝类中存在的一些特有基因家族和扩张基因家族参与了其适应较为复杂的埋栖生活环境。高质量青蛤全基因组图谱的完成,为青蛤抗逆与生长发育等重要性状的遗传解析及良种选育提供了重要的理论依据和科学基础。
《Cell》是与《Nature》、《Science》并列的国际三大顶级期刊之一。《iScience》是《Cell》出版的新创综合性期刊,主要发表包括生命科学、物理学、地球学以及环境科学在内的创新性基础和应用研究。
文章链接:https://www.sciencedirect.com/science/article/pii/S2589004220303333